設計と実装
このページでは、BMTKとBioNetの背景、およびbionetliteの設計思想と実装の詳細について説明します。
BMTKについて
BMTK (Brain Modeling Toolkit) は、神経科学コミュニティのためのモデリング・シミュレーションフレームワークです。
モジュラー設計
BMTKは、意図的に以下の3つのモジュールに分割されています:
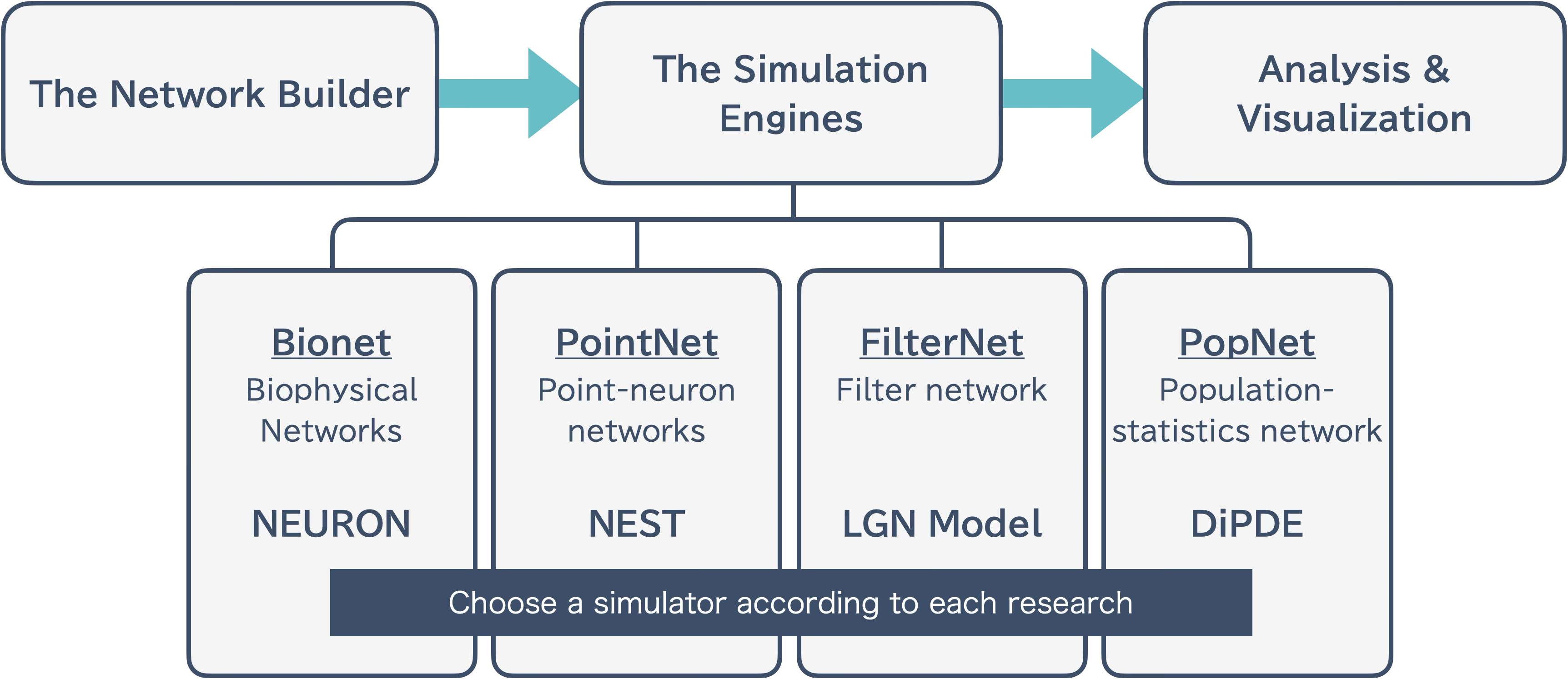
The Network Builder: ネットワーク構築
The Simulation Engines: シミュレーション実行
Analysis & Visualization: 結果の解析と可視化
この分離により、大規模なシミュレーションの実行が容易になります。
データ標準化
BMTKは、神経科学コミュニティのデータ標準化形式に対応しています:
SONATA: ネットワーク記述形式
Allen Cell Types Database: モデルパラメータのデータベース
BioNetについて
BioNetは、BMTKのモジュールの1つで、生物物理学的ニューロンネットワークを扱います。
BioNet Builder
ネットワークの構築部分だけを担当するモジュールです。
ユーザはネットワークにニューロンやシナプスを追加するコードを記述
実行すると、H5ファイルやCSVファイルなどの形でネットワーク情報が出力される
bionet simulatorは、これらのファイルを用いて実行を行う
大規模なネットワークではネットワークの構築だけでも計算コストが高く、シミュレータ部分と独立して動作することがエラーハンドリングの観点などから有用です。
使用例:
from bmtk.builder.networks import NetworkBuilder
# Create network
net = NetworkBuilder('mcortex')
# Add nodes
net.add_nodes(...)
# Add edges
net.add_edges(...)
# Build and save
net.build()
net.save_nodes(output_dir='network')
net.save_edges(output_dir='network')
BioNet Simulator
bionet builderが生成したファイルを読み込み、NEURON シミュレータを用いてシミュレーションを実行します。
bionetliteの設計
bionetliteは、bionetの拡張として設計されています。既存コードの最低限の変更で、Neuliteカーネル用のファイルを生成します。
NetworkBuilderのオーバーライド
bionetliteは、bmtk.builder.networksのNetworkBuilderを拡張します:
# bionet case
from bmtk.builder.networks import NetworkBuilder
# bionetlite case
from bionetlite import NeuliteBuilder as NetworkBuilder
このシンプルなimportの変更により、以下が実現されます:
bionetの全ての機能がそのまま使用可能
NeuliteBuilder固有の処理が追加される
ユーザコードの変更は最小限
処理フロー
bionetliteの処理フロー:
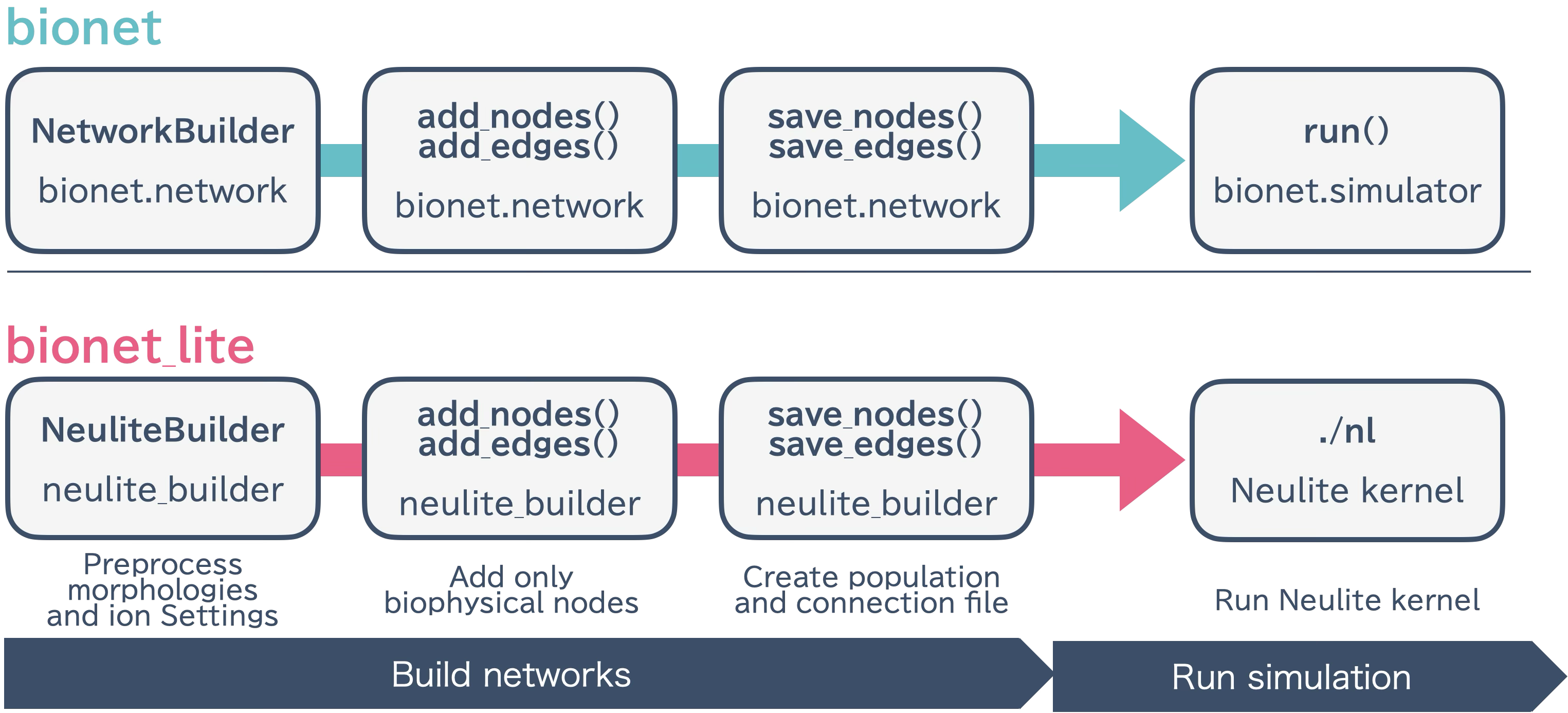
NeuliteBuilderの初期化 - 形態ファイルとイオンチャネル設定の前処理を設定
add_nodes() / add_edges() - bionetの処理をそのまま実行し、biophysicalノードのみを記録
save_nodes() - bionetの処理に加えて、
<network_name>_population.csvを生成save_edges() - bionetの処理に加えて、シナプス接続位置を決定し
<src>_<trg>_connection.csvを生成
並列実行のサポート
bionetliteは、MPI並列実行をサポートしています:
# Parallel execution with 4 processes
mpirun -n 4 python build_network.py
並列実行時の処理:
各MPIプロセスが独立してネットワーク構築を実行
ファイル出力は適切に同期される
大規模ネットワークの構築時間を大幅に短縮