Tutorial 1: 単一細胞の定常電流入力
概要
このチュートリアルでは、単一の生物物理学的ニューロンに対して定常電流入力を行う最もシンプルな例を示します。
参考
対応するBMTKチュートリアル: Tutorial: Single Cell Simulation with Current Injection
bionetからbionetliteへの変更
変更が必要なのはimport文だけです。 それ以外のコードは完全に同じです。
変更前(BMTK):
from bmtk.builder.networks import NetworkBuilder
変更後(bionetlite):
from bionetlite import NeuliteBuilder as NetworkBuilder
コード例
from bionetlite import NeuliteBuilder as NetworkBuilder
# Create network
net = NetworkBuilder('mcortex')
# Add a single biophysical neuron
net.add_nodes(
N=1,
pop_name='Scnn1a',
model_type='biophysical',
model_template='ctdb:Biophys1.hoc',
dynamics_params='472363762_fit.json',
morphology='Scnn1a_473845048_m.swc'
)
# Build the network
net.build()
# Save to files
net.save_nodes(output_dir='sim_ch01/network')
生成されるファイル
このシミュレーションでは以下のファイルが生成されます:
<network_name>_population.csv
ポピュレーション情報を含むファイル。単一のニューロンの生物物理学的特性を定義します。
id,pop_name,model_type,morphology,dynamics_params,...
0,Scnn1a,biophysical,Scnn1a_473845048_m.swc,472363762_fit.json,...
<src>_<trg>_connection.csv
シナプス接続情報を含むファイル。このチュートリアルではシナプス接続は定義されていないため、ファイルの中身は空です(ヘッダーのみ)。
注釈
Neuliteカーネルの実行のためにはconnectionファイルが必要です。接続が存在しない場合、空のファイルが生成されます。
処理済みswcファイル
Neulite用に前処理された形態ファイルが生成されます。
このファイルは Perisomatic model に従って処理されており、以下の変更が加えられています:
実際の軸索形態を簡略化された人工軸索に置き換え
ゼロベースのインデックスへの変換
深さ優先探索によるソート
注釈
Perisomatic modelの詳細については、仕様と制限事項 を参照してください。
Neuliteが生成するファイル
bionetliteは以下のディレクトリ内にファイルを生成します:
スクリプトに
base_dirが設定されている場合:{base_dir}_nl/base_dirが設定されていない場合:neulite/
ディレクトリ構造
sim_ch01_nl/
├── mcortex_population.csv
├── mcortex_mcortex_connection.csv
├── kernel/
│ └── config.h
└── data/
├── Scnn1a_473845048_m.swc
└── 472363762_fit.csv
mcortex_population.csv
Neulite用のポピュレーションファイル。カラム構成は以下の通りです:
#n_cell,n_comp,name,swc_file,ion_file
1,3682,default_100_100,data/Scnn1a_473845048_m.swc,data/472363762_fit.csv
#n_cell: Number of cellsn_comp: Number of compartmentsname: Population nameswc_file: Path to SWC morphology fileion_file: Path to ion channel parameter file
mcortex_mcortex_connection.csv
Neulite用の接続ファイル。このチュートリアルでは接続がないため、ヘッダーのみのファイルが生成されます:
#pre nid,post nid,post cid,weight,tau_decay,tau_rise,erev,delay,e/i
kernel/config.h
Neuliteカーネル用のコンパイル時設定ファイル。BMTKのconfig.jsonから自動生成されます:
// Automatically generated by bionetlite
#pragma once
// Simulation parameters
#define TSTOP ( 2000.0 )
#define DT ( 0.1 )
#define INV_DT ( ( int ) ( 1.0 / ( DT ) ) )
// Neuron parameters
#define SPIKE_THRESHOLD ( -15.0 )
#define ALLACTIVE ( 0 )
// Current injection parameters
#define I_AMP ( 0.12 )
#define I_DELAY ( 500.0 )
#define I_DURATION ( 1000.0 )
このファイルには以下のパラメータが含まれます:
TSTOP: シミュレーション時間(ms)DT: タイムステップ(ms)INV_DT: 1ミリ秒あたりのステップ数SPIKE_THRESHOLD: スパイク検出閾値(mV)I_AMP: 電流注入の振幅(nA)I_DELAY: 電流注入開始時刻(ms)I_DURATION: 電流注入の持続時間(ms)
注釈
config.hはNeuliteカーネルのビルド時に使用されるため、このファイルを変更した場合はカーネルの再ビルドが必要です。
Neuliteカーネルのビルドと実行
ステップ1: カーネルソースの展開
Neuliteのkernelディレクトリの内容をbionetliteが生成したkernelディレクトリにコピーします。config.hはbionetliteが生成済みなので除外します:
rsync -av --exclude='config.h' /path/to/neulite/kernel/ sim_ch01_nl/kernel/
ステップ2: カーネルのビルド
kernelディレクトリに移動してビルドします。config.hは既にbionetliteによって生成されています:
cd sim_ch01_nl/kernel
make
ステップ3: バイナリの配置
ビルドしたバイナリファイル nl を1階層上にコピーします:
cp nl ../
ステップ4: シミュレーションの実行
bionetliteが生成したディレクトリに移動して、シミュレーションを実行します:
cd ..
./nl mcortex_population.csv mcortex_mcortex_connection.csv
シミュレーション結果ファイル
Neuliteカーネル実行後、以下のテキストファイルが生成されます:
s.dat - スパイク時刻データ
スペース区切りのテキスト形式で、各行に1つのスパイクイベントを記録:
Time(ms) NeuronID
125.3 0
156.8 0
189.2 0
1列目: スパイクが発生した時刻(ms)
2列目: スパイクを発生させたニューロンのID
v.dat - 膜電位データ
スペース区切りのテキスト形式で、各行に全ニューロンの膜電位を記録:
Time(ms) Neuron0_Vm Neuron1_Vm ...
0.0 -65.0 -65.0
0.1 -64.8 -64.9
0.2 -64.6 -64.8
1列目: 時刻(ms)
2列目以降: 各ニューロンのソーマ膜電位(mV)
注釈
これらのファイルはテキスト形式(ASCII)なので、Pythonのnumpyやpandasで簡単に読み込めます。
BMTKとの実行結果の比較
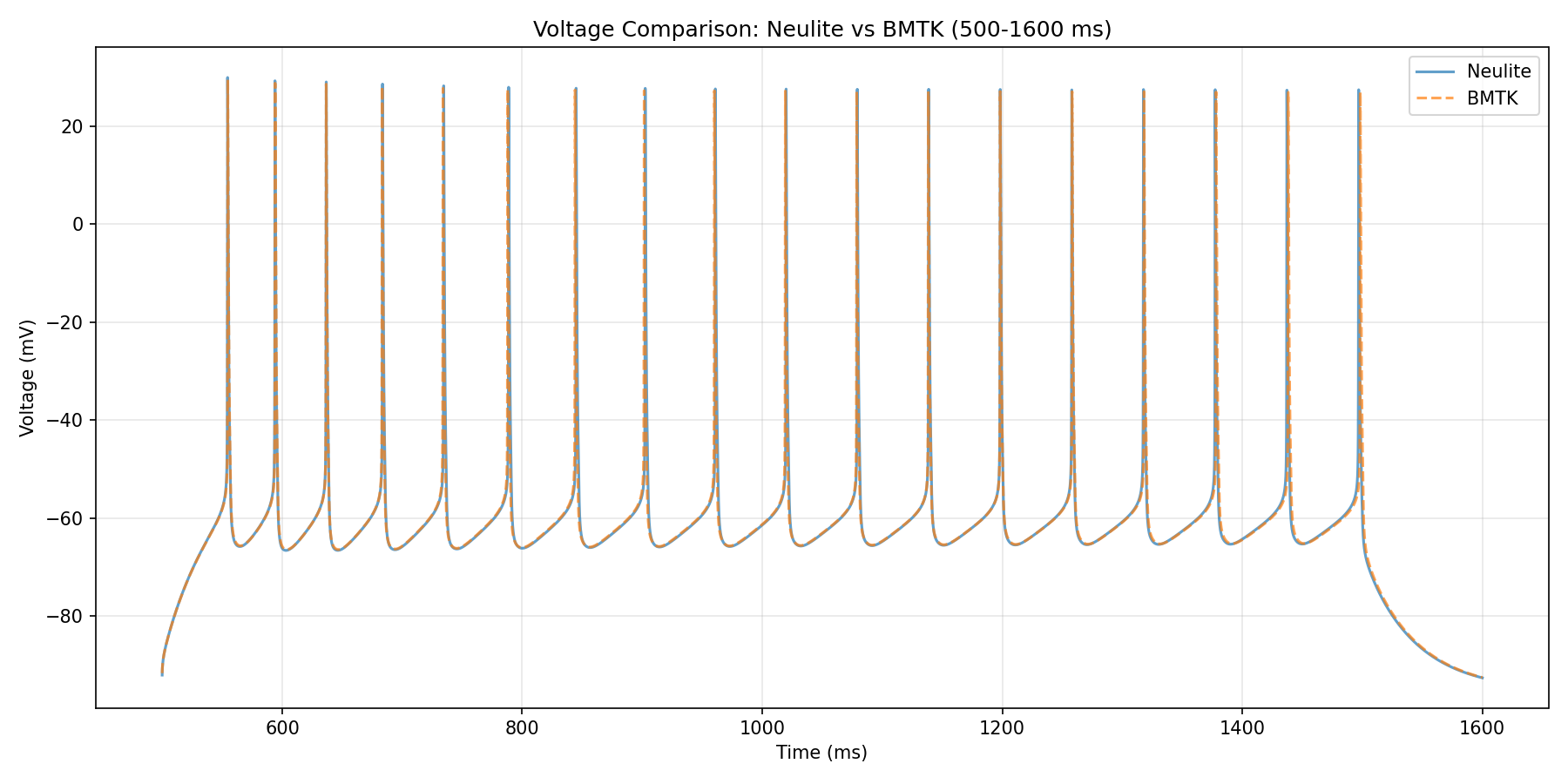
次のステップ
Tutorial 2: スパイク入力シミュレーション - スパイク入力を使用したシミュレーション
基本的な使い方 - より詳細な使用方法
注釈
このチュートリアルの完全なコード例とデータファイルは、リポジトリのexamplesディレクトリにあります。